À la découverte de notre ADN
L'objectif de ce TP est de dmettre en pratique le langage python pour construire et analyser un brin d'ADN. L'idée est, dans un premier temps, de reprendre les éléments de base du langage (condition, boucles ...) pour créer des fonctions qui construise un brin d'ADN, le lisent, réalise une transcription une traduction ... puis d'écrire une classe qui réalise ces actions.
Introduction
Ce n'est pas l'objet de faire ici un cours complet sur l'ADN. Vous trouverez de nombreuses choses sur le sujet. On va juste rappeler quelques éléments de base pour qu'un non biologiste puisse faire le TP.
ADN ?
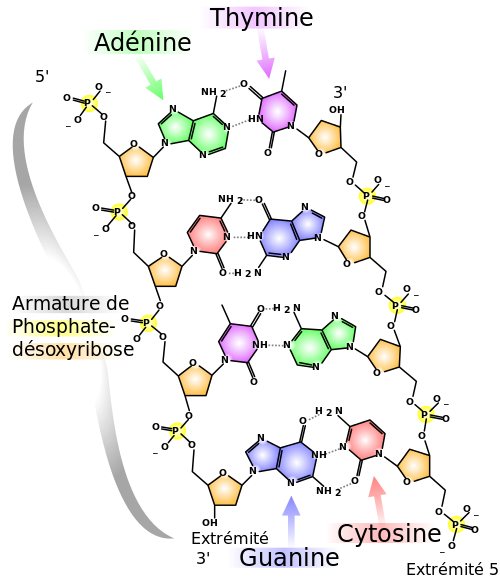
L'ADN, pour Acide DésoxyriboNucléique, est une macromolécule constituée de deux brins qui forme une double hélice maintenue par des liaisons hydrogène. Ces brins sont formés par un enchainement de maillons appelés, nucléotides qui contiennent les bases de l'ADN :
- A pour Adénine
- T pour Thymine
- G pour Guanine
- C pour Cytosine
Les bases de l'ADN fonctionnent par paire, une sur chaque brin : adénine avec thymine et guanine avec cytosine.
Traduction et transcription
La transcription est un mécanisme qui permet de "recopier" l'ADN dans le noyau de la cellule pour former un ARN (acide ribonucléique) qui sera utilisé dans la cellule notamment lors de la traduction. L'ARN présente la même structure que l'ADN mais lors de la transicription, la thymine (T) est remplacé par l'uracile (U).
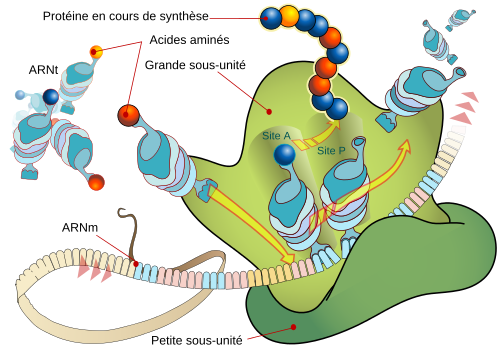
La traduction de l'ADN consiste à lire l'ARN issue de la transcription pour synthétiser une protéine avec l'aide de la machinerie cellulaire. L'ARN est découpé en codons qui sont constitués de 3 bases et correspondent à un acide aminé, c'est le code génétique. Les codons sont lus les uns à la suite des autres et la protéines est assemblée comme une chaîne (peptidique) d'acides aminés.
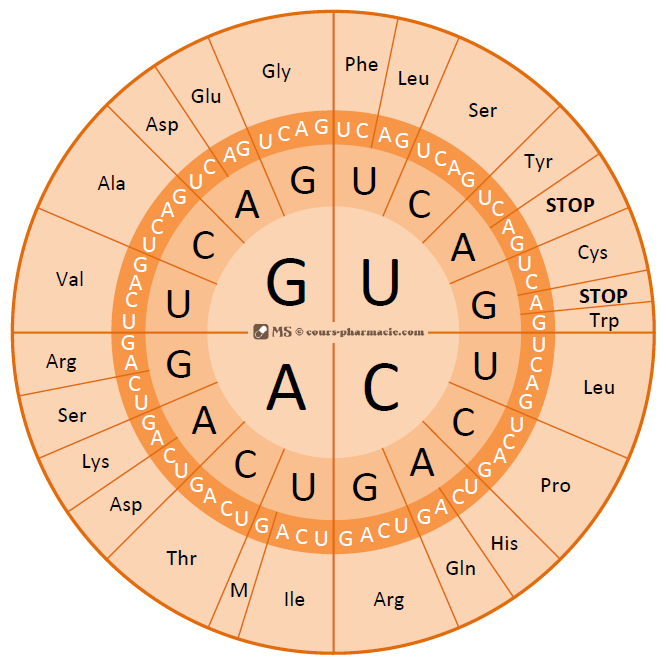
Correspondance codons - acides aminés
Le schéma ci-dessous vous donne la correspondance entre un codon, composé de trois bases de l'ARN et un acide aminé.
Par exemple, GUA est un codon qui code pour l'acide aminé Val, c'est à dire la Valine. On remarquera que plusieurs codons peuvent coder pour le même acide aminé ce qui limite la portée des erreurs de copies ou des mutations. On note également la présence de codons STOP indiquant la fin de la partie "codante" et qui stoppe la synthèse de la protéine.
Les dictionnaires python
Pour entrer le code génétique dans python on pourra utiliser les dictionnaires. Ces objets python sont un peu comme des listes ils contiennent plusieurs autres objets. À la différence des listes, les éléments d'un dictionnaire sont repérés par une clef et non par un indice. On peut utiliser tout type de clef : des nombres, des chaînes de caractères ou même des tuples.
Petit aperçu
>>> aa = dict()
>>> aa
{}
Une liste est délimitée par des crochets, un dictionnaire par des accolades. Par contre, comme pour les listes, La clef est donnée entre crochets :
>>> aa["M"] = "Met"
>>> aa["L"] = "Leu"
>>> aa["A"] = "Ala"
>>> print("dictionnaire : ", aa)
>>> print("un élément : ", aa["L"])
dictionnaire : {'A': 'Ala', 'L': 'Leu', 'M': 'Met'}
un élément : Leu
Remarque : Les dictionnaires ne sont pas ordonnés. On voit sur l'exemple ci-dessus que bien que la clef "A" ait été ajoutée en dernier, elle apparait en premier dans le dictionnaire.
On peut lister les clefs et les valeurs d'un dictionnaire ou les deux et
les parcourir avec une boucle for.
Liste des clefs :
>>> print(aa.keys())
>>> for key in aa:
>>> print(key)
dict_keys(['A', 'L', 'M'])
A
L
M
Pour les valeurs :
print(aa.values())
for val in aa.values():
print(val)
dict_values(['Ala', 'Leu', 'Met'])
Ala
Leu
Met
On peut aussi parcourir les deux simultanément :
print(aa.items())
for key, val in aa.items():
print(key, " = ", val)
dict_items([('A', 'Ala'), ('L', 'Leu'), ('M', 'Met')])
A = Ala
L = Leu
M = Met
aa.items() retourne une liste de tuple de la forme (clef, valeur).
Petit exercice
- Construire un dictionnaire qui met en relation le code à une lettre et le code à trois lettre des acides aminés. Voir ce tableau.
- Construire un dictionnaire qui pour un acide aminé donné donne plusieurs
informations :
* Code à 1 lettre, * Code à 3 lettres, * Polarité * Masse * pI * ...
Conseil : Les dictionnaires sont des objets permettant de structurer des données. C'est un modèle simpliste de base de données. Il est tout à fait possible de les imbriquer.
Pour le code génétique
Pour le code génétique on peut envisager plusieurs solutions :
- la clef est un tuple correspondant au codon :
>>> gencode = {("A", "U", "A"): "Ile", ("U", "G", "A"): "STOP"}
>>> print(gencode)
{('U', 'G', 'A'): 'STOP', ('A', 'U', 'A'): 'Ile'}
- la clef est un acide aminé, la valeur est la liste des codons associés
>>> gencode = {"Phe": ["UUU", "UUC"], "Met": ["AUG"]}
>>> print(gencode)
{'Met': ['AUG'], 'Phe': ['UUU', 'UUC']}
Questions
L'idée est d'écrire dans un premier temps des fonctions qui réalisent les opérations suivantes puis d'écrire une classe qui contient les même fonctionnalités.
- Écrire une fonction qui génère aléatoirement un brin d'ADN. On pourra choisir aléatoirement un codon STOP pour terminer le brin ou la partie codante.
- Écrire une fonction qui écrit le brin d'ADN dans un fichier
- Écrire une fonction qui lit un brin d'ADN dans un fichier
- Identifier s'il s'agit d'un brin d'ADN ou d'ARN et si ce brin est valide
- Statistique : extraire les informations suivantes d'un brin d'ADN
- Nombre total de bases
- Nombre de codons
- pourcentage de chaque base dans le brin
- Écrire une fonction qui réalise la transcription de l'ADN en ARN
- Écrire une fonction qui traduit l'ARN et renvoie la chaîne d'acides aminés correspondante. Attention, elle doit s'arrêter au codon STOP.
- Statistique. Extraire des statistiques sur les acides aminés
- Le nombre d'acides aminés
- Le pourcentage d'acide aminé polaire
- Le nombre de chaque acide aminé différent
Programmation
À vous de jouer !
Commencez par vous exercer sur les dictionnaires avec l'exercice proposé puis vous pourrez transcrire et traduire votre ADN. Introspection garantie, à la fin du travail vous vous connaitrez sur le bout des doigts !
La correction de ce problème est disponnible sous la forme d'un notebook jupyter.